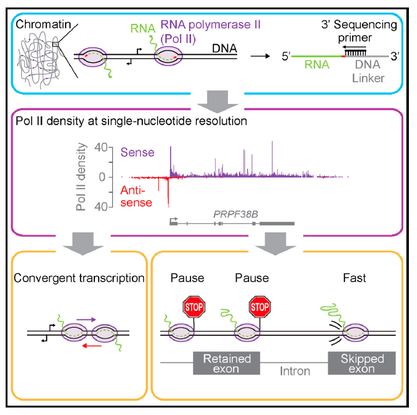
Τι μπόρεσαν να δουν όμως οι Mayer και συνεργάτες στο συγκεκριμένο άρθρο (που βρέθηκε στο εξώφυλλο του Cell τον περασμένο Απρίλιο); Τα βασικά τους ευρήματα συνοψίζονται σε δύο πολύ πρωτότυπες παρατηρήσεις. Την "συγκλίνουσα μεταγραφή" (convergent transcription) και τη ρύθμιση του splicing στο μεταγραφικό επίπεδο.
Η πρώτη παρατήρηση είναι τόσο ενδιαφέρουσα όσο και αναπάντεχη. Μελετώντας το σήμα επιμηκούμενων πολυμερασών γύρω από σημεία έναρξης της μεταγραφής πρωτεϊνικών γονιδίων, οι ερευνητές κατέγραψαν ένα έντονο σήμα στο συμπληρωματικό κλώνο για τα πρώτα 150-200 νουκλεοτίδια καθοδικά του σημείου έναρξης για το 25% των γονιδίων. Από παλιότερες δουλειές γνωρίζαμε ήδη από τα τέλη της προηγούμενης δεκαετίας την ύπαρξης της "αποκλίνουσας μεταγραφής" (divergent) κατά την οποία anti-sense μετάγραφα παράγονται από το σημείο έναρξης της μεταγραφής και ανοδικά του. Η ανακάλυψη των καθοδικών anti-sense μεταγράφων αποτελεί πραγματικό αίνιγμα για τους μοριακούς βιολόγους. Γιατί να συμβαίνει; Είναι άραγε μια απλή "παρενέργεια" του ανοίγματος της χρωματίνης κατά την έναρξη της μεταγραφής και της "εκκίνησης" των σταθμευμένων ως τότε πολυμερασών ή έχει μια συγκεκριμένη λειτουργία; Οι ερευνητές συσχετίζουν τα 25% περίπου γονίδια με συγκλίνοντα μετάγραφα με τη χαμηλή τους έκφραση και προτείνουν ότι η σύγκρουση δύο πολυμερασών που κινούνται η μία έναντι της άλλης θα μπορούσε να λειτουργήσει σαν κατασταλτικός μηχανισμός. Γιατί όμως το κύτταρο να "επιλέξει" κάτι τόσο πολύπλοκο; Τι απογίνονται τα μετάγραφα που προκύπτουν; Τα ερωτήματα που προκύπτουν είναι όπως συνήθως περισσότερα από τις απαντήσεις.
Το δεύτερο σημαντικό εύρημα της συγκεκριμένης δουλειάς είναι η σύνδεση των επιμηκούμενων πολυμερασών και του μηχανισμού του splicing. Αναλύοντας έναν μεγάλο αριθμό από μετάγραφα με πολλές εναλλακτικές μορφές, οι ερευνητές παρατήρησαν σημαντικές διαφορές στο σήμα επιμήκυνσης γύρω από διαφορετικούς τύπους εξονίων. Ενώ στα συσταστικά εξόνια (σε αυτά που ενσωματώνονται σε όλες τις πιθανές ισομορφές ενός γονιδίου κατά το splicing) και στα εναλλακτικά εξόνια που τελικώς χρησιμοποιούνται, η πολυμεράση "φρενάρει" και δείχνει να καθυστερεί, το αντίθετο συμβαίνει στα εξόνια τα οποία τελικά δεν ενσωματώνονται σε ώριμα μετάγραφα, πάνω από τα οποία η πολυμεράση φαίνεται να περνά χωρίς να "σταματήσει". Τι μας λέει αυτό το εύρημα; Ότι κατά πάσα πιθανότητα η πολυμεράση αναγνωρίζει τα εξόνια με κάποιο τρόπο και ακόμα πιο σημαντικά δίνει χρόνο σε άλλους μηχανισμούς που ενδεχομένως σχετίζονται με το splicing να "αποφασίσουν" για την πορεία των εξονίων. Δίνει μάλιστα περισσότερο χρόνο στα εξόνια που τελικά ενσωματώνονται σε ώριμα μετάγραφα και λιγότερο στα άλλα. Κι εδώ τα ερώτηματα που προκύπτουν δεν είναι λίγα. Ποια είναι τα "σήματα" που αναγνωρίζει η πολυμεράση ώστε να "φρενάρει" στα "σωστά" εξόνια; Και γιατί εφόσον σταματά στο 5' άκρο τους κάνει το ίδιο αφού διατρέξει το σώμα των εξονίων και στο 3';
Τόσο η συγκλίνουσα μεταγραφή, όσο και σύζευξη με το splicing αναδεικνύουν τον ρόλο που διαδραματίζει η χρωματίνη και οι εναλλακτικές της διαμορφώσεις. Στα σημεία έναρξης της μεταγραφής, τα γονίδια που εμφανίζουν συγκλίνουσα μεταγραφή έχουν χαρακτηριστικές "δίρροπες" κατανομές νουκλεοσωμάτων που διαφέρουν από τα γονίδια στα οποία έχουμε αποκλίνουσα ή μονο-κατευθυνόμενη μεταγραφή. Αφετέρου, για την περίπτωση της μεταγραφής γύρω από εξόνια, η σύνδεση με τη δομή της χρωματίνης είχε ήδη γίνει από το 2009 με εργασίες (μεταξύ άλλων) και από το δικό μας group, όταν παρατηρήσαμε ότι εξόνια που πρόκειται να ενσωματωθούν σε ώριμα μετάγραφα έχουν πολύ διαφορετικό (και εντονότερο) σήμα κατάληψης από νουκλεοσώματα. Στενά προσδεδεμένα νουκλεοσώματα θα μπορούσαν να λειτουργήσουν σαν "φράγματα" για την πολυμεράση, καθυστερώντας την και δίνοντας έτσι περισσότερο χρόνο στον μηχανισμό αναγνώρισης των εξονίων να τα "επισημάνει" προς ωρίμανση.
Τι περιμένουμε από εδώ και περά; Περισσότερη προσπάθεια για τον καθορισμό αυτών των χρωματινικών υπογραφών για τη ρύθμιση αυτών των διαδικασιών. Μια πρώτη σύνδεση της αποκλινουσας μεταγραφής με πρότυπα χρωματινικής διαμόρφωσης δημοσιεύθηκε πρόσφατα. Αλλά περισσότερα για αυτά τα θέματα σε επόμενα post.
 RSS Feed
RSS Feed
